Möglicher Ansatz für neue Antibiotika entdeckt
Zwei Forschungsteams aus Bern und Zürich haben eine neue Methode entwickelt, um einen Einblick in einen bislang kaum erforschten Teil der bakteriellen Proteinherstellung zu erhalten. Die Resultate könnten bei der Entwicklung neuer Antibiotika helfen.
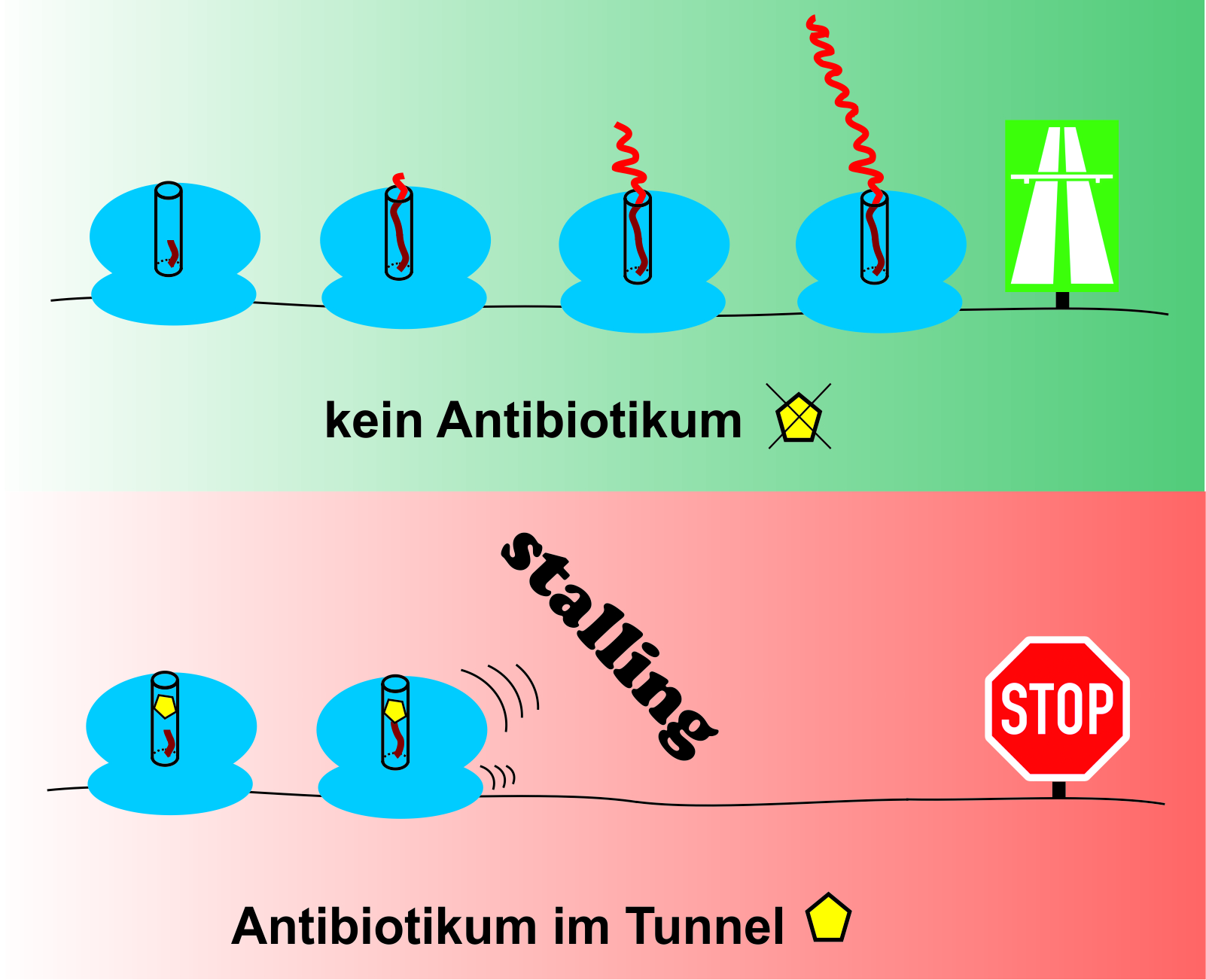
Ribosomen sind die «Fabriken» der Zelle und für die Produktion von Proteinen zuständig. Sie entsprechen einer komplexen Maschinerie aus ribosomalen RNA-Molekülen und diversen ribosomalen Proteinen. So lange diese Fabriken funktionieren, werden tief im Zentrum des Molekülkomplexes unablässig Aminosäuren zu Proteinketten zusammengesetzt, die sich allmählich durch den sogenannten Ribosomal Exit Tunnel herauswinden. Manchmal allerdings kommt das «Montageband» ins Stocken. Die Proteine bleiben im Exit-Tunnel stecken und die ganze Maschinerie wird blockiert.
Der Vorgang, mit dem englischen «Stalling» bezeichnet, wird schnell bedrohlich für die Zelle, wenn viele Ribosomen betroffen sind. Aus diesem Grund ist Stalling eine effiziente Strategie von Antibiotika, um Bakterien-Zellen abzutöten. Über die Hälfte der natürlich vorkommenden Antibiotika zielt auf die Ribosomen ab. Ein vielversprechender Weg zur Entwicklung neuer Wirkstoffe führt deshalb über die Analyse des Mechanismus, wo und wie genau diese Antibiotika die Proteinfabrik zum Stillstand bringen. Dies ist nötig, um die weltweit zunehmenden multiresistenten Keime zu bekämpfen, die sich aus einem breiten und häufig unangemessenen Einsatz von Antibiotika bei Mensch und Tier ergeben. «Es braucht daher einen gezielten Einsatz von neuen Antibiotika, um diese Resistenzen umgehen zu können», sagt Prof. Norbert Polacek vom Departement Chemie und Biochemie der Universität Bern und Gruppenleiter im Nationalen Forschungsschwerpunkt «RNA & Disease». Gemeinsam mit der Gruppe von Prof. Jonathan Hall vom Departement für Chemie und angewandte Biowissenschaften der ETH Zürich hat er nun einen möglichen Ansatz für neue Antibiotika entdeckt. Die Studie wurde im Fachjournal «Nucleic Acids Research» publiziert.
«Bremsvorgang» beschrieben
Das Stalling ist ein aussichtsreicher Startpunkt für diese Forschung, da die Funktion des Ribosomal Exit Tunnels bei der Polypeptidsynthese und der folgenden Faltung der Proteine erst in den Grundzügen bekannt ist. Der Tunnel scheint so etwas wie ein Bremspedal für den Translationsprozess zu sein. Manchmal verlangsamt sich durch die Blockade der Prozess nur – was womöglich auch Vorteile bei der Verfertigung der Proteine mit sich bringt –, manchmal sorgen spezifische Interaktionen der frischgebackenen Proteine mit der Tunnelwand aber auch für einen Totalhalt. Wie genau geht dieser Stopp vonstatten? Und wie merken die Stationen weiter unten in der Fabrikationslinie, dass sie die Arbeit ebenfalls einstellen müssen?
Die beiden Forschergruppen untersuchten dazu das Stalling, welches von Erythromycin und anderen sogenannten Makrolidantibiotika – welche die Proteinsynthese von Bakterien hemmen – verursacht wird. Sie entwickelten eine Methode, um kleinste Bestandteile des Tunnels zu verändern und zu untersuchen, welchen Effekt das auf das Stalling hatte. Indem sie einzelne Nukleobasen oder sogar nur einzelne Atome in der ribosomalen RNA austauschten, konnten sie die Rolle spezifischer funktionaler Gruppen im Tunnel beim Stalling aufzeigen. Sie konnten auch die exakten Teile der Ribosom-Maschinerie identifizieren, die für das Übermitteln des Stalling-Signals vom Tunnel zurück zum Peptidyl-Transferase-Zentrum des Ribosoms verantwortlich sind, wo die Aminosäuren zu Proteinen zusammengehängt werden und wo der Betrieb dann auch gestoppt wird. Diese Nukleobasen tragen direkt zwar nicht viel zum Mechanismus der Proteinsynthese bei, doch erklärt vermutlich diese besondere Rolle bei der Überwachung der wachsenden Peptidstränge im Exit-Tunnel, warum sie im Lauf der Evolution weitgehend unverändert erhalten blieben.
Vereinte Kräfte für komplexe Probleme
Die Erkenntnisse sind nicht nur für die Medikamentenforschung von Interesse, sondern auch, weil sie das Potenzial interdisziplinärer Ansätze bei komplexen molekularbiologischen Problemen aufzeigen. «Die Arbeit hätte von keiner unserer beiden Gruppen allein bewältigt werden können», sagt Norbert Polacek. Beide Gruppen brachten ihre spezifische Expertise ein, die Hall-Gruppe von der ETH das Knowhow bei der chemischen Synthese der RNA und die Polacek-Gruppe von der Uni Bern die Biochemie-Kenntnisse des Ribosoms. Laut Polacek sei diese Forschungsarbeit ein perfektes Beispiel der Philosophie des Nationalen Forschungsschwerpunkts ‹RNA & Disease – Die Rolle von RNS in Krankheitsmechanismen›, der verschiedene Disziplinen zusammenbringt, um das Verständnis der Wechselwirkungen zwischen der RNA und dem Organismus bei Krankheiten zu vertiefen.
Publikationsangaben:Miriam Koch, Jessica Willi, Ugo Pradère, Jonathan Hall and Norbert Polacek: Critical 23S rRNA interactions for macrolide-dependent ribosome stalling on the ErmCL nascent peptide chain, Nucleic Acids Research, 2017, 21. März, doi: 10.1093/nar/gkx195 |
10.05.2017